16. Los Genomas como Textos Antiguos, Parte 2
14 de Junio de 2013. Temas: Genética
Nota: Esta serie de artículos ha sido concebida como una introducción básica a la ciencia de la evolución para no especialistas. Aquí se puede ver la introducción a esta serie o volver al índice aquí.
En este artículo examinamos las secuencias de ADN de varias especies de mosca de la fruta para probar la hipótesis de que todas ellas surgieron por procesos de especiación.
En el artículo anterior discutíamos algunas características que podríamos esperar al comparar genomas entre especies similares, en el caso de que éstas descendieran de una población ancestral común. Volviendo a nuestra analogía del “libro” con los genomas, destacábamos que lo primero en que tendríamos que fijarnos sería en la estructura general de los dos genomas propuestos como descendientes de un genoma antecesor común: ¿Están los “capítulos”, los “párrafos”, etc. en el mismo orden? ¿Utilizan las mismas “frases”?, y así sucesivamente. En otras palabras, ¿parece que los genomas de las especies existentes son copias ligeramente modificadas unas de otras?
La respuesta a esta cuestión, por parte de la moderna genómica comparada, es un enfático sí. Lo que vemos al comparar los genomas de las especies que sospechamos emparentadas es que, en gran parte, parecen copias los unos de los otros. En algunos casos, la coincidencia entre los genomas puede ser de más del 95%, monómero a monómero del ADN, para el conjunto de los dos genomas. No sólo tienen las dos los mismos genes, sino que los tienen en el mismo orden en sus cromosomas, teniendo cada cromosoma de una especie su correspondencia en la otra. Imaginemos encontrarnos un libro que sea idéntico a otro manuscrito en el 95%, con los capítulos, los párrafos y las frases todos en el mismo orden, con sólo pequeñas diferencias entre ellos: esta es la impresión que uno tiene al comparar los genomas entre algunas especies.
Un grupo que ha sido analizado con cierto detalle son las diversas especies de mosca de la fruta, del género Drosophila. Los científicos ya han determinado las secuencias del genoma completo de doce de las especies de Drosophila, y los han comparado entre sí. Algunas especies tienen genomas casi idénticos, exactamente lo que uno podría predecir si hubieran sido, alguna vez, la misma especie con un genoma común. Aunque no es posible mostrar aquí grandes cantidades de secuencias de ADN, veamos un pequeño fragmento de una “frase”, es decir de un gen, en tres especies de mosca de la fruta (Drosophila yakuba, Drosophila simulans y Drosophila sechellia), todas ellas consideradas como especies distintas de moscas:
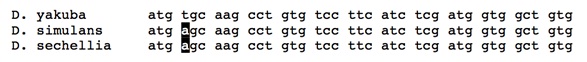
La primera impresión que uno tiene, al revisar las secuencias, es que son casi idénticas. Esto no resulta extraordinario en estas tres especies; de hecho, este patrón se aplica casi a cada uno de los genes que tienen, y las tres tienen los mismos genes. Lo segundo que uno percibe es que, sin embargo, hay algunas raras diferencias: en este pequeño fragmento, dos de las especies (D. simulans y D. sechellia) tienen una “a” en la cuarta posición de este gen, donde D. yakuba tiene una “t”.
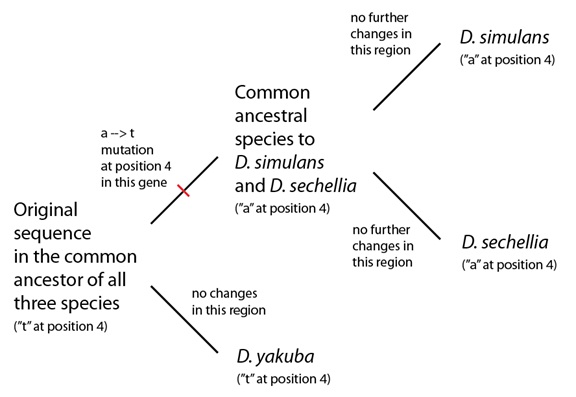
Volvamos a pensar en la analogía que utilizábamos en el artículo anterior: la de las ediciones del libro y los errores tipográficos compartidos entre ellas. Estas secuencias, en estas tres especies, son muy parecidas a las hipotéticas ediciones de nuestra analogía y podemos interpretar el patrón que observamos de una forma muy parecida:
simulans(“a” en la posición 4)
no más cambios en esta zona
Especie antecesora común de
D. simulans y D. sechellia
(“a” en la posición 4)
no más cambios en esta zona
D. sechellia(“a” en la posición 4)
a - t mutación
en la posición 4
de este gen
Secuencia
original en el
antecesor común
a las tres especies sin cambios
en esta zona
D. yakuba (“t” en la posición 4)
(Los lectores sagaces notarán también que la otra posibilidad es que la “a” de la cuarta posición provenga del texto original, que la “t” sea el error tipográfico, y que haya tenido lugar, en algún momento, una mutación (de t a a), en el linaje que conduce a D. yakuba. Si estamos considerando esta opción: bien hecho. El truco para decidir cuál es el texto original consiste en revisar tantas copias como sea posible; y en este caso, cuando revisamos un gran número de especies de Drosophila, vemos una “t” en la cuarta posición en la mayoría de las otras especies, y una “a” sólo en D. simulans y D. sechellia. Esto significa que la elección más razonable es que la “t” sea el original, y la “a” sea una mutación).
Aunque esto es sólo un pequeño ejemplo, ilustra lo que los científicos observan al comparar los genomas de las especies que piensan que están relacionadas, basándose en otros criterios, como la morfología. Lo que ven es precisamente lo que uno esperaría si, en efecto, las especies en cuestión hubieran sido formadas por procesos de especiación: genomas casi idénticos, con pequeños cambios compartidos entre algunas de las especies.
Identidad más allá de lo necesario para la funcionalidad a nivel del ADN
Otra observación más, que apoya la hipótesis de que estas secuencias son copias de una secuencia ancestral, es que el nivel de identidad entre ellas, de coincidencia en la secuencia, es incluso mayor de lo que se requeriría, incluso cuando se considera la función del gen. Volvamos al fragmento de gen que estábamos considerando. Esta secuencia, como principio de un gen, codifica una proteína con la misma función en las tres especies. (Si se necesita refrescar cómo los genes están constituidos por los monómeros de ADN, que finalmente se traducen en una secuencia de monómeros de aminoácidos, podemos ver los dos artículos anteriores de esta serie, los titulados “El Fundamento de la Variación Heredable, parte 1, y parte 2”). En estas especies, esta proteína tiene, en los ocho primeros monómeros (aminoácidos), la siguiente secuencia:

Aminoácidos: Metionina, (cisteína o serina), lisina,
prolina, valina, serina, fenilalanina,
isoleucina, serina
Como puede verse, el segundo aminoácido es diferente en las dos secuencias, pero los demás aminoácidos son idénticos. Lo importante ahora es darse cuenta de que hay muchas, muchas maneras de escribir esta “frase” y llegar al mismo significado (la secuencia de aminoácidos). Esto es posible porque, para la mayoría de los aminoácidos, hay varias combinaciones de monómeros de ADN (de tres nucleótidos) que producen el mismo aminoácido al ser traducidas. Por ejemplo, la secuencia de D. yakuba podría escribirse, entre otras muchas opciones, también así:

Esta secuencia es bastante diferente de la que vemos en las otras dos especies:

En este caso, sólo coinciden 14 de los 27 monómeros de ADN: una identidad de sólo un 52%. Sin embargo, lo que observamos entre estas especies, es que coinciden 26 de los 27 monómeros: más del 96% de identidad. En otras palabras, sería posible que estos dos genes fueran mucho menos parecidos, a nivel del ADN, y aún tener las mismas secuencias de aminoácidos que observamos en las dos especies. Además, lo que vemos al comparar los dos genes, es que coinciden a nivel de ADN mucho más de lo que en realidad tendrían que parecerse para llegar a tener la misma secuencia de aminoácidos. Una explicación simple es que las dos secuencias coinciden porque han sido copiadas de una misma secuencia original.
Identidad más allá de lo necesario para la funcionalidad a nivel de aminoácidos
Una segunda observación que apoya la hipótesis de que las secuencias génicas de D. yakuba, D. simulans y D. sechellia, son en realidad copias, proviene de la comparación con otras especies de moscas menos estrechamente relacionadas con estas tres. Todas las especies de Drosophila examinadas hasta ahora tienen este gen, pero en especies más alejadas la secuencia puede ser algo distinta. Por ejemplo, este gen en D. mojavensis tiene la siguiente secuencia de ADN:

De nuevo, parte de la secuencia permanece idéntica en las cuatro especies (apoyando la hipótesis de que también esta última secuencia es una copia, aunque con más cambios) pero ahora las diferencias que vemos son mayores. A pesar de estas diferencias, la versión del gen de D. mojavensis es, sin embargo, perfectamente funcional, y realiza exactamente el mismo trabajo que en las otras tres especies, que sí estaban más estrechamente relacionadas.
Así que, lo que estas observaciones indican es que no hay necesidad biológica de que los genes sean casi idénticos a nivel de aminoácidos, ni siquiera a nivel de ADN, en las distintas especies. Numerosas secuencias de aminoácidos, e incluso numerosas secuencias de ADN, son igualmente capaces de realizar la misma función. Así que, lo que vemos una y otra vez, (¡ a lo largo de todos los genomas!), son genes casi idénticos, con sólo unas pocas diferencias que, a menudo, incluso son compartidas; exactamente lo que sería esperable que produjera la especiación.
¿Y qué hay del “diseño común”? 1
Una pregunta que me suelen hacer, cuando presento este tipo de datos, es la del “diseño común” como explicación alternativa. En otras palabras, ¿Podrían estos tipos de modelos explicarse como especies creadas separadamente, que no compartieran un antecesor común, sino que hubieran sido diseñadas, y creadas, para tener los mismos genes o similares, porque esos genes tienen que tener las mismas o similares funciones?
Ya hemos visto los problemas básicos de esta línea argumental: que los genes, y los genomas enteros, de especies similares coinciden mucho más de lo que debieran, y que las diferencias que observamos, en especies estrechamente relacionadas, están dispuestas en el mismo orden que uno predeciría si se hubieran formado por especiación.
Tan solo simples moscas
Desde luego, a la mayoría de los cristianos no les quita el sueño la posibilidad de que numerosas especies de moscas de la fruta surgieran, a lo largo del tiempo, a través de múltiples procesos de especiación. Como hemos mencionado antes, incluso los Creacionistas de la Nueva Tierra aceptan sucesos de especiación como esos. Lo que resulta más polémico, desde luego, es la cuestión de si ese modelo del antecesor compartido incluye a nuestra propia especie. En el artículo siguiente de esta serie examinaremos esta cuestión, comparando el genoma humano con los genomas de nuestros parientes vivos supuestamente más cercanos: los grandes simios o monos superiores.
Notas
—Nota del T.: En el original: “common design”


