18. Los Genomas como Textos Antiguos, Parte 4
28 de Junio de 2013. Temas: Genética
Nota: Esta serie de artículos ha sido concebida como una introducción básica a la ciencia de la evolución para no especialistas. Aquí se puede ver la introducción a esta serie o volver al índice aquí.
En este artículo comparamos las secuencias génicas entre la especie humana y otros mamíferos, para comprobar la hipótesis de que son copias modificadas derivadas de un genoma ancestral.
En el artículo anterior veíamos que a una escala de organización amplia, el genoma humano tiene las características que debe tener si, como sucede, compartimos un antecesor común con otros homínidos (“grandes simios”). Siguiendo con nuestra analogía del “libro”, vamos ahora a comparar estos textos a una menor escala, de mayor detalle: la de las frases y las palabras.
Comparación de los genomas al nivel de las “frases”
En un artículo anterior comparábamos la secuencia de ADN de un gen encontrado en una serie de especies de Drosophila. Estas comparaciones son también posibles utilizando secuencias del ADN de mamíferos, incluidos los seres humanos y otros primates, y el patrón que observamos resulta ahora familiar:

Como veíamos en las secuencias de Drosophila, este gen es prácticamente idéntico en una serie de especies. Más específicamente, la secuencia humana y las secuencias de otros tres primates (chimpancés, gorilas y orangutanes) difieren sólo en un puñado de monómeros de ADN: como mucho, 4 de 90 son diferentes. Tampoco hay, como ya hemos visto, necesidad biológica de que estas secuencias sean idénticas; de hecho, incluso para esta pequeña región de este gen, hay más de 53 millones de formas diferentes de codificar exactamente la misma secuencia de aminoácidos. La mayoría de estas secuencias tienen muchas más diferencias con la secuencia humana que las secuencias casi idénticas que observamos en otros primates. Por ir algo más allá, tampoco hay ninguna necesidad biológica en particular para que este gen contenga exactamente la misma secuencia de aminoácidos que vemos compartida entre los primates. En otros organismos, como los perros y los lobos por ejemplo, una secuencia ligeramente diferente realiza la misma función igual de bien.
Desde luego, no se pueden mostrar franjas grandes de secuencias ADN en este formato. Sin embargo, este pequeño segmento de un gen es representativo de los genes, e incluso de genomas enteros, entre los primates. La comparación detallada de todas las secuencias génicas entre humanos y chimpancés, por ejemplo, revela que son en un 99.4% idénticos, en una muestra total de 1.85 x 107 (18 millones) de monómeros de ADN. Hay que destacar que las regiones del genoma que codifican para genes son una pequeña minoría de secuencias del genoma; humanos y chimpancés tienen más de 3.0 x 109 (3 mil millones) de monómeros de ADN en sus genomas. De estos 3 mil millones de monómeros, 2700 millones presentan, al alinearlos, sólo una diferencia del 1,23 % entre ellos.
Es decir, que al comparar las secuencias del ADN entre humanos y otros primates, vemos exactamente el patrón que hubiéramos predicho basándonos en un antecesor común; un patrón consistente con pequeñas modificaciones de un genoma ancestral.
Buscar errores tipográficos
En un artículo anterior de esta serie discutíamos cómo la replicación del ADN es un proceso muy preciso, pero no perfecto. Estas dos características de la replicación del ADN hacen que las mutaciones puedan afectar a los genes en el momento de copiarse y que las copias que se hagan en el futuro, a partir de genes mutados, transmitan fielmente la mutación de ahí en adelante (al menos hasta que vuelva a suceder una nueva mutación en ese mismo punto, que vuelva a cambiar las cosas). Esto significa que las secuencias génicas pueden persistir en los genomas durante mucho tiempo después de haber mutado en genes no funcionales si no existe una desventaja selectiva por el hecho de haber perdido esa función. Si una mutación produce una desventaja, la selección natural tenderá a eliminarla de la población, como ya hemos discutido anteriormente.
Un ejemplo de esto es el gen que codifica para una enzima (L-gulonolactona oxidasa, o “GULO”) que es necesaria para la síntesis de vitamina C en los mamíferos. La mayoría de los mamíferos sintetizan su propia vitamina C a partir de otros componentes de la dieta, y el gen GULO es necesario para el último paso en ese proceso que convierte un precursor de la vitamina C en el producto final. Como hemos visto en otros genes, la secuencia de este gen se conserva entre las distintas especies de mamíferos- que tiene una secuencia casi idéntica que se mantiene mediante la selección natural. Por ejemplo, una parte de este gen en vacas, perros y ratas tiene la secuencia siguiente (con las diferencias con respecto a la secuencia de la vaca marcadas en negro):

Bos taurus Vaca
Canis lupus Perro / Lobo
Rattus norvegicus Rata
En estas tres especies el gen es funcional, por supuesto, y las tres pueden sintetizar su propia vitamina C sin obtenerla directamente de sus dietas.
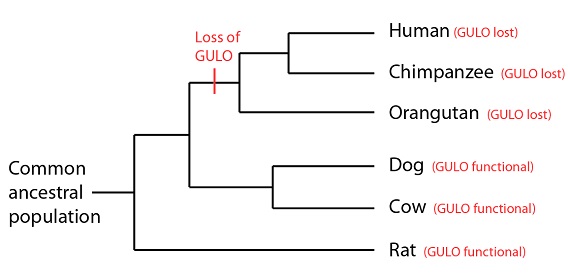
La especie humana, desde luego, no puede sintetizar su vitamina C; padecemos el escorbuto si no la obtenemos a través de la dieta. Esta situación, atípica para un mamífero, la compartimos con otros homínidos (“grandes simios”), y por la misma razón. Aunque estas especies tienen parte de la secuencia de ADN del gen GULO, tiene tantas mutaciones en él que vuelven al gen incapaz de sintetizar una enzima funcional. La misma región del gen GULO, mostrada en la figura anterior, tiene la misma secuencia en humanos, chimpancés y orangutanes (mostrando en negro, ahora, las diferencias con la secuencia humana):

Homo sapiens Hombre
Pan troglodytes Chimpancé
Pongo pygmaeus Orangután
Bos taurus Vaca
Canis lupus Perro / lobo
Rattus norvegicus Rata
Secuencias funcionales del gen GULO de vaca, perro, y rata comparadas con las secuencias no funcionales en varios primates. Se muestra una porción del exon 12. Se señalan en negro las diferencias con la secuencia humana. Los primates comparten una única deleción de un nucleótido en esta región resaltada en amarillo.
Una vez más observamos que las secuencias de los primates son casi idénticas entre sí. Sin embargo, aquí vemos algo nuevo, que es que estas tres copias del gen GULO son no funcionales, en parte debido a una mutación por deleción; la eliminación de un monómero de ADN (marcada en amarillo en las secuencias de los primates). Esta deleción es idéntica en las tres especies, poniendo en evidencia que es un “error tipográfico compartido” copiado de un texto previo- o, en términos biológicos, una mutación por deleción que tuvo lugar una sola vez en el antecesor común de humanos, chimpancés y orangutanes, y que se ha heredado luego en las tres especies. Los perros, las vacas y las ratas, sin embargo, se ramificaron aparte del linaje que lleva a los primates antes de que esa deleción sucediera:
Pérdida Hombre (GULO perdido)
de GULO Chimpancé (GULO perdido)
Población Orangután (GULO perdido)
ancestral
común Perro (GULO funcional)
Vaca (GULO funcional)
Rata (GULO funcional)
La pérdida de la función del GULO no parece haber supuesto una desventaja selectiva para los primates de aquel momento; probablemente porque tenían una dieta rica en vitamina C. De hecho, incluso en los humanos, esa pérdida no es un problema serio hasta que uno se encuentra sin una fuente de vitamina C durante un período de tiempo prolongado.
La nariz sabe
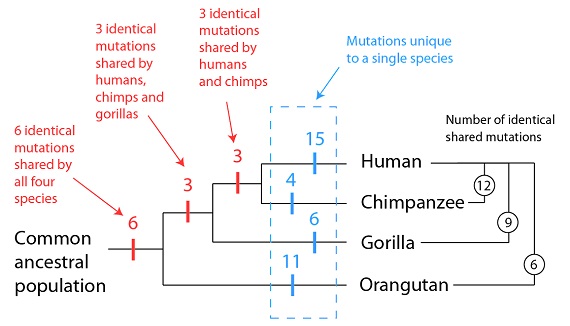
Hay otro ejemplo tan interesante como el del GULO, y es un ejemplo que ya he examinado con más detalle en otro contexto [http://biologos.org/blog/a-tale-of-three-creationists-part-3]. No es más que otro de los muchos ejemplos de mutaciones idénticas compartidas entre el genoma humano y el de otros primates. Un estudio que trataba, con detalle, sobre mutaciones compartidas entre los primates se refería a los genes relacionados con el sentido del olfato. Estos genes codifican los llamados receptores olfativos que son proteínas que se encuentran en la membrana de las células del epitelio nasal de los mamíferos. Los receptores olfativos funcionan mediante la adhesión de los compuestos que hay en el aire, cambiando de forma en el proceso, y señalando ese cambio de forma al sistema nervioso, en lo que nosotros percibimos como sentido del olfato. La acción combinada de numerosos receptores olfativos, actuando conjuntamente, es lo que confiere a un determinado olor sus características distintivas. Los mamíferos dedican una parte desproporcionada de su genoma a los genes de sus receptores olfativos, muy probablemente por lo útiles que dichos genes resultan para encontrar comida, encontrar pareja y, en general, para la percepción de su propio ambiente. A pesar de su utilidad, estos genes pueden también mutar y perderse y, de hecho, el genoma humano demuestra que nuestra especie ha perdido varios por mutación. Sin embargo, como en el caso del gen GULO, estas secuencias defectuosas de genes olfativos persisten en una forma reconocible. Pero, lo que nos importa más para nuestro propósito aquí es, en cambio, el patrón que estos genes mutados forman al compararlos con los genomas de otros primates. Como introdujimos ya, con nuestra analogía de los libros copiados, esperamos encontrar algunos errores tipográficos compartidos entre los textos, y otros que sean únicos en una edición. En los genes olfativos defectuosos observamos precisamente estas dos categorías: mutaciones compartidas y mutaciones únicas:
6 mutaciones 3 mutaciones 3 mutaciones
idénticas idénticas idénticas
compartidas compartidas compartidas Mutaciones únicas
por las 4 por humanos, por humanos y en una sola especie
especies chimpancés chimpancés
y gorilas
Número de mutaciones Hombre idénticas compartidas
Población Chimpancé
ancestral Gorila
común Orangután
Como podemos ver en el diagrama anterior, los humanos comparten el máximo de mutaciones idénticas de los genes olfativos con los chimpancés, algo menos con los gorilas, y todavía menos con los orangutanes. De las 12 mutaciones que son idénticas entre los humanos y los chimpancés, 9 son idénticas también con los gorilas, y 6 lo son con los orangutanes. Estas mutaciones compartidas, y el patrón que encontramos en ellas, se explican fácilmente a través de un antecesor común. Las mutaciones que son únicas en una de las especies también se explican fácilmente como surgidas después de que las poblaciones se separaran (en azul).
También es importante destacar lo que no vemos, al comparar estas mutaciones entre distintas especies de primates. No observamos mutaciones idénticas entre humanos y gorilas, por ejemplo, a no ser que veamos siempre también exactamente la misma mutación en los chimpancés. Esto concuerda perfectamente si la población antecesora común de humanos y gorilas lo fuera también de humanos, gorilas y chimpancés. De la misma manera, si observamos idénticas mutaciones compartidas entre humanos y orangutanes podemos predecir con confianza que observaremos esas mismas mutaciones en los gorilas y en los chimpancés, como de hecho sucede. Este patrón de mutaciones compartidas es precisamente lo que uno predeciría si, de hecho, ello fuera el resultado de un antecesor común, sin que nada quede sin encajar.
Múltiples evidencias, una única conclusión
En el artículo siguiente de esta serie, cerraremos el círculo y discutiremos cómo las múltiples líneas de evidencias genómicas de la evolución humana que hemos examinado, son coherentes en un modelo o patrón en el que se apoyan unas a otras.


