22. Introducción a la Homoplasia y a la Convergencia Evolutiva
2 de Agosto de 2013. Temas: Genética, Historia de la Vida
Nota: Esta serie de artículos ha sido concebida como una introducción básica a la ciencia de la evolución para no especialistas. Aquí se puede ver la introducción a esta serie o volver al índice aquí.
En este artículo examinamos cómo la evidencia de la repetición de sucesos evolutivos puede encontrarse, precisamente, buscando en una filogenia las características que no concuerden con un árbol de especies.
En los últimos artículos de esta serie introducíamos el concepto de que las características individuales, tales como las secuencias génicas individuales, pueden no siempre coincidir con la filogenia o árbol filogenético de especies de un grupo de organismos relacionados. La clasificación del linaje incompleta es una de las formas en que esto puede suceder; otra manera es que surjan similitudes a través de sucesos independientes. Tales características tendrían superficialmente una apariencia de haber sido heredadas de un antecesor común pero, en realidad, serían ejemplos de homoplasias: características compartidas entre especies, pero que no han sido heredadas de un antecesor común.
Tal para cual1
Un ejemplo clásico de homoplasia es el vuelo activo de las aves y de algunos mamíferos como los murciélagos. El árbol filogenético de especies de aves, murciélagos y mamíferos no voladores (por ejemplo ratones) sitúa a todos los mamíferos juntos, como más estrechamente relacionados entre sí que cualquiera de ellos con las aves. Entonces, para explicar el carácter compartido del vuelo en murciélagos y aves, tenemos que considerarlo como una homoplasia; como algo independiente que surgió en dos linajes separados:
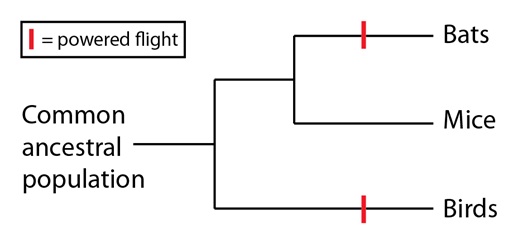
La explicación alternativa de que el vuelo activo fuera homólogo entre los murciélagos y las aves, y que por tanto estuviera presente ya en su pasado antecesor común, requeriría que todos los mamíferos, salvo los murciélagos, hubieran perdido la capacidad de volar (por no decir nada de la cantidad de información de secuencias de ADN que apoyan el árbol filogenético de especies anterior). Mas allá de esta evidencia, también hay una buena razón procedente de la anatomía comparada para pensar que el vuelo activo surgió independientemente en murciélagos y aves. Para lograr la sustentación, las aves utilizan las plumas sujetas a lo largo de sus extremidades anteriores. En cambio, los murciélagos, para formar sus alas, utilizan una membrana que une entre sí los dedos, y éstos con el cuerpo:

Fuentes de las imágenes: Murciélago | Ave
Ambas soluciones funcionan bien; pero cuando analizamos en detalle el rasgo del “vuelo activo” y estudiamos sus componentes, vemos que, aunque el carácter en conjunto es un carácter convergente, los componentes que lo hacen posible no lo son. Esta observación apoya fuertemente la conclusión de que el vuelo activo surgió de forma separada en aves y mamíferos.
Homoplasia versus Homología
Podemos ilustrar un ejemplo de cómo aparece la homoplasia en una simple secuencia de ADN, utilizando una filogenia. Supongamos que tres especies tienen la secuencia siguiente en una parte de un mismo gen:

Basándonos sólo en estos datos, la filogenia más simple, la más económica, sería la siguiente:
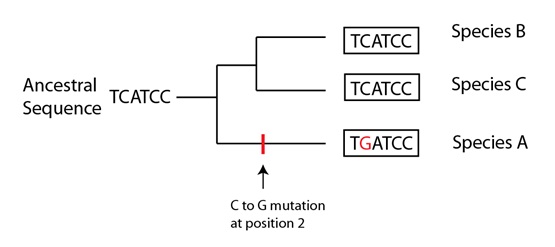
Basándonos en estos datos, la secuencia ancestral que inferiríamos sería “TCATCC”, y la rama de la filogenia que conduce hasta la Especie A habría tenido una mutación para explicar la diferencia que observamos en la secuencia. En ausencia de otras evidencias, esta filogenia sería la que mejor encajaría con los datos.
Este cuadro tan claro, sin embargo, podría ser contradicho por nuevos datos; datos que demuestran que el sencillo árbol de especies que hemos construido arriba es, de hecho, incorrecto. Y si lo es, tendríamos que encajar esas secuencias en un árbol filogenético de especies diferente, lo cual significa que tendremos que explicar el patrón planteando más de una mutación. Veamos un ejemplo hipotético para mostrar el proceso.
Supongamos que comparamos los datos de las secuencias de varios cientos de genes más, de estas tres especies, así como los de un cierto número de otras especies relacionadas que no aparecen en nuestro árbol de especies. Supongamos también que estos datos apoyan firmemente un árbol filogenético de especies diferente al que habíamos generado (en la amplísima mayoría de los casos los datos apoyan un árbol con las Especies A y B como más cercanamente emparentadas, y con la Especie C como más lejanamente relacionada). Esto nos “obligaría” a redibujar el árbol de especies de la manera siguiente, poniendo las secuencias cortas originales en un patrón diferente, con sus especies:
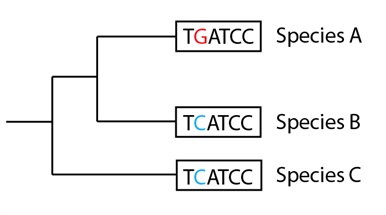
Y supongamos, además, que los datos de la secuencia de ADN de este gen en particular, procedentes de una especie adicional no incluida en el árbol de especies, indicaran que la secuencia ancestral en realidad tenía una “T” en esa segunda posición, en lugar de una “C”:
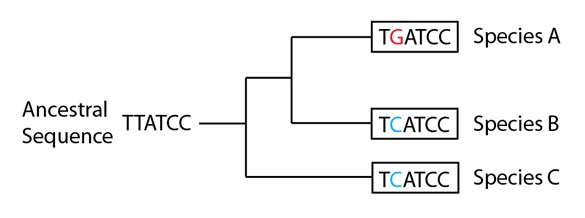
Ahora tenemos que explicar que las tres especies de nuestro árbol tengan una secuencia que no es la ancestral, en la segunda posición, y además intentar que tengan sentido las mutaciones que llevaron hasta el patrón que vemos ahora. Recordemos que todavía seguimos constreñidos a buscar la explicación más económica para el conjunto de los datos sin embargo, para este gen en particular, nos vemos obligados a aplicar múltiples mutaciones para que sea adecuado el patrón en el árbol de especies. Aún así elegimos esa opción, sin embargo, porque sería todavía más improbable para múltiples mutaciones que el patrón de cientos de otras secuencias génicas hubiera sido moldeado de una forma coordinada; y que dichas secuencias apoyen esta versión del árbol de especies.
Si nos tomamos un tiempo para tratar de “resolver” el árbol de genes añadiendo mutaciones al árbol de especies, enseguida nos daremos cuenta de que al menos se necesitan tres mutaciones para producir el patrón observado. Hay, desde luego, soluciones con más de tres mutaciones pero resultan explicaciones menos probables. Aquí abajo mostramos una de esas posibles soluciones:
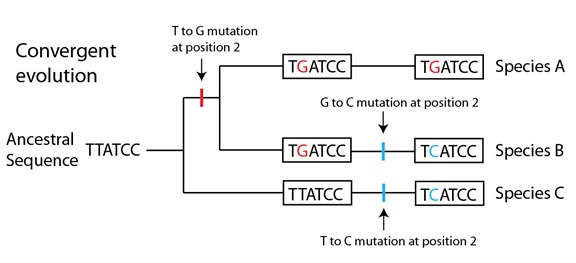
En la rama de la filogenia que conduce a las Especies A y B, hay una mutación de T a G anterior a la divergencia A / B, representada por la barra roja. Otra mutación, la segunda, aparece luego en el linaje que lleva a la Especie B, que cambia la G de esa misma posición por una C, representada por una barra azul. Independientemente, el linaje que lleva a la Especie C tiene también una mutación en esa misma posición, cambiando la T ancestral por una C, también representada por una barra azul. El resultado final es que dos de las secuencias, en las Especies B y C, han llegado a ser idénticas, pero ninguna de ellas heredó la “C” de la segunda posición de su antecesor común. En otras palabras, han llegado al mismo “destino final” desde distintos puntos de partida: han “convergido” en una misma secuencia. No sorprende, pues, que este fenómeno se conozca como evolución convergente o convergencia evolutiva. En estas dos especies, la “C” de la segunda posición no es homóloga, la similitud no se debe a haber sido heredada de un antecesor común sino más bien a una homoplasia, una similitud resultante de sucesos independientes en los dos linajes.
Las homoplasias pueden ser tan simples como un único cambio en un monómero de ADN, como en este ejemplo, o tan complejas como la reorganización independiente de sistemas múltiples, con numerosos genes y partes del cuerpo, que finalmente convergen en una solución, como era el caso del vuelo activo de aves y murciélagos. Sin embargo, en ambos casos podemos determinar que surgieron como sucesos independientes, en linajes separados, porque esas características no encajan en los árboles de especies como sucesos únicos.
El poder de la convergencia
Como las homoplasias funcionan como marcadores que señalan sucesos evolutivos repetidos, el buscar homoplasias en árboles de especies es una manera útil de testar las hipótesis sobre la reproducibilidad de la evolución; de ver con qué frecuencia las especies convergen en soluciones similares. Al final, parece que la evolución es notablemente repetible en multitud de rasgos generales. Hay numerosos ejemplos de innovaciones independientes repetidas a lo largo de la historia evolutiva, algunas de las cuales serán examinadas con más detalle en próximos artículos:
- Forma corporal hidrodinámica: la forma hidrodinámica de formas acuáticas de vida, tales como los peces, los ictiosaurios, las ballenas, las focas o las aves buceadoras como los pingüinos, son todas independientes, adaptaciones convergentes al modo de vida acuático.
- Vuelo activo: además de las aves y los murciélagos, el vuelo activo evolucionó también independientemente en los insectos y los pterodáctilos.
- Ecolocación: algunos mamíferos, como los murciélagos o las ballenas, han desarrollado independientemente sistemas que les permiten localizar el alimento detectando los ecos de las ondas, producidas por ellos mismos y que, reflejadas, proceden de los objetos o de las presas de su medio ambiente.
- Ojos en cámara: la evolución repetida de los ojos en cámara, es decir, ojos que utilizan una lente, es uno de los ejemplos más llamativos de convergencia evolutiva. Los ojos en cámara han evolucionado independientemente en cnidarios (algunas medusas), cefalópodos (como el calamar y el pulpo) y vertebrados (aves, mamíferos).
Un hecho destacable es que estos ejemplos generalizados de convergencia son todos moldeados por el medio ambiente físico de los organismos en cuestión; la percepción de la luz (los ojos), la capacidad de volar por el aire (las alas), o la de moverse eficientemente por el agua (cuerpos hidrodinámicos). La presencia constante de esas características ambientales hacía previsible que moldearan la adaptación de muchas especies.
Ahondando más
En el siguiente artículo de esta serie, miraremos con más detalle algunos ejemplos de evolución convergente a nivel molecular y veremos evidencias de que, en algunos casos, la homología de hecho se encuentra escondida subyacente bajo algunos sucesos convergentes.
Notas
- Nota del T.: En el original ““Birds of a feather…”. El dicho completo en inglés es: “Birds of a feather… flock together”, que de manera figura se puede traducir al castellano por “Dios los cría y ellos se juntan” o bien “Tal para cual” que es la expresión completa que se ha adoptado.


